Investigador: Dra Susana Correa García.
Instituto: Departamento de Química Biologica, Facultad de ciencias Exactas y Naturales, Universidad de Buenos Aires.
jueves, 23 de octubre de 2008
Decimoséptima semana (16/10/08)
Preparamos dos tubos, uno con H2O, Buffer, vector, inserto y la ligasa, y el otro igual pero sin el inserto, esto va a servir para poder verificar que las dos puntas del vector no se hayan unido de vuelta. Lo dejamos en un baño de agua a 26ºC durante una hora para que se ligue correctamente el inserto con el vector. Luego procedimos con el siguiente protocolo:
Transformación de E. coli
1. Poner 100 ml de células competentes y 20 ml de rx de ligación en un eppendorff estéril. Hacer control con 100 ml de células competentes y 0.5 ml de plásmido control (purificado x kit QUIAGEN)
2. Incubar 30 minutos en hielo
3. Incubar 2 minutos a 42 ˚C
4. Incubar 5 minutos en hielo
5. Agregar 1 ml (o 450 ml) de medio LB sin Ampicilina
6. Incubar 1 hora a 37 ˚C a 225 rpm
7. Centrifugar las células y resuspender el pellet en el sobrenadante restante
8. Sembrar todo el contenido de cada tubo en placas con medio LB + AmpicilinaColocar en estufa a 37 ˚C
miércoles, 15 de octubre de 2008
Decimosexta semana (02/10/08)
Preparamos un gel de agarosa donde pusimos los insertos y dejamos correr.
miércoles, 1 de octubre de 2008
Decimoquinta semana (25/09/08)
Procedimos de igual manera;
crecimos las cepas en placas de medio rico,limpiamos, centrifugamos, usamos una solución que nos ayuda a que el plásmido entre en la célula, incubamos y plaqueamos.
Decimocuarta semana (18/09/08)
 Observamos los resultados de la anterior semana.
Observamos los resultados de la anterior semana.Usamos el plásmido que purificamos en una de las semanas y lo cortamos con una enzima de restricción (HindIII). Lo hicimos por duplicado.
Esto se llama hacer una digestión de un plásmido. Se usa un buffer específico, las enzimas, el plásmido y se rellena con agua. Luego se coloca a 37°C durante el periodo de una hora y media.
En un gel de agarosa 8% m/v corrimos el plásmido sin digerir en diferentes concentraciones y los dos plásmidos digeridos también en diferentes concentraciones.
miércoles, 17 de septiembre de 2008
Decimotercera Semana (04/09/08)
Para llevar a cabo todo esto:
-crecimos las cepas en placas de medio rico
-resuspendimos células en 1 ml de agua estéril y centrifugamos
-descartamos el sobrenadante y resuspendimos en el liquido remanente
-agregamos una solución que nos ayuda a que el plásmido entre en la célula (sumado al hitshock del siguiente paso)
-de la suspensión celular pasamos 20microl a otro eppendorf y le agregamos el ADN plasmidico
-los incubamos 30 minutos a 42°
-agregamos 1ml de agua esteril, centrifugamos para bajar las células
-y plaqueamos en el medio
domingo, 17 de agosto de 2008
Introducción
Cada célula posee en su interior un núcleo que contiene, entre otras cosas, la información (ADN) para que ésta pueda funcionar debidamente. Esta información a su vez, se subdivide en pequeños segmentos, en paquetes que son llamados genes. Cada gen, luego de ser transcripto y traducido, produce una proteína en especial; es decir, cada gen tiene la suficiente información para poder sintetizar a una proteína.
 Como modelo experimental, se utilizarán células de la levadura Saccharomyces cerevisiae que es un organismo eucariota unicelular no patógeno, de fácil manipulación, de rápido crecimiento en medios de cultivos definidos y cuya secuencia genómica completa es conocida. Teniendo en cuenta la conservación evolutiva de las vías de señalización disparadas en respuesta a los nutrientes del medio, el uso de S. cerevisiae como modelo para el estudio de transducción de señales permitirá esclarecer el gran desafío que es conocer cómo señales producidas por nutrientes presentes en el medio extracelular se integran y son coordinadas por las células.
Como modelo experimental, se utilizarán células de la levadura Saccharomyces cerevisiae que es un organismo eucariota unicelular no patógeno, de fácil manipulación, de rápido crecimiento en medios de cultivos definidos y cuya secuencia genómica completa es conocida. Teniendo en cuenta la conservación evolutiva de las vías de señalización disparadas en respuesta a los nutrientes del medio, el uso de S. cerevisiae como modelo para el estudio de transducción de señales permitirá esclarecer el gran desafío que es conocer cómo señales producidas por nutrientes presentes en el medio extracelular se integran y son coordinadas por las células. En esta imágen se puede ver una célula de levadura gemando (reproduciéndose asexualmente).
En esta imágen se puede ver una célula de levadura gemando (reproduciéndose asexualmente).Se puede observar tambien el núcleo y su vacuola.
UGA4 es el gen que codifica para la proteína Uga4. Esta proteína es una permeasa que se encuentra en la membrana plasmática y permite la incorporación de los ácidos d-aminolevúlico (ALA)3,4 y y-aminobutírico (GABA)5,6.


El GABA puede ser utilizado por las células de levadura como fuente de nitrógeno, pero es una fuente pobre, en comparación con fuentes ricas de nitrógeno como amonio. Hay factores que influyen en la represión o en la inducción de la expresión del gen, dependiendo de la situación en la que la célula se encuentre. El gen se va a expresar sólo en ausencia de una fuente rica en nitrógeno (como amonio); en ausencia de este tipo de fuente, la célula desencadena mecanismos para poder sobrevivir en dichas condiciones. En la secuencia promotora del gen UGA4 hay dos sitios importantes conocidos como UAS-GABA y UAS-GATA. En cada uno de ellos influyen factores que al pegarse reprimen o inducen su expresión. La expresión del gen UGA4 depende de la presencia de un inductor, GABA5,7. La inducción requiere la acción de dos factores de transcripción, uno específico, Uga3, y otro pleiotrópico, Uga35, que actúan a través de una secuencia activadora rica en los nucleótidos GC (UASGABA)8,9. La región regulatoria del gen UGA4 contiene también cuatro repeticiones adyacentes del heptanucleótido 5´-CGAT(A/T)AG-3´ que constituye un elemento UASGATA. Sobre este último actúan los factores GATA que pueden actuar como factores positivos y negativos. Los factores negativos (Uga43 y Gzf3) compiten con los positivos (Gln3 y Gat1) y el que “gana” se pega al promotor del gen. Cuando hay una fuente rica de nitrógeno presente, la célula no gasta energía sintetizando proteínas que transporten GABA8,10,11, los factores positivos. Gln3 y Gat1, se encuentran en el citoplasma unidos a la proteína Ure2, no pueden actuar sobre el elemento UAS-GATA, y por ende, los factores negativos “ganan”, lo que lleva a la represión de la expresión de UGA4. En este caso los factores negativos se unen al sitio UAS-GATA.

Cuando no hay una fuente rica pero sí hay GABA en el medio, la célula se adapta al cambio y activa toda la maquinaria para sintetizar la proteína Uga4; en otras palabras, GABA induce la expresión del gen UGA45,7,9. Los factores positivos se disocian de Ure2 y se translocan al núcleo donde desplazan a los factores negativos uniéndose al promotor y permitiendo la expresión del gen9,12. La interacción entre los factores positivos Gln3 y Gat1 con Ure2 está modulada por el estado de fosforilación de estos factores que a su vez depende de la actividad kinasa de Tor13-15.
El objetivo principal de esta pasantía es la familiarización con distintas técnicas de biología molecular mediante la participación en un proyecto en el cual se estudia la cascada de señales que lleva a la regulación de la expresión del gen UGA4 por aminoácidos extracelulares. Los mecanismos moleculares en dicha cascada de señales no se conocen por completo pero se sabe que el complejo sensor SPS está involucrado.
Este complejo se conoce como SPS16-18 por sus tres componentes proteicos (Ssy1p, Ptr3p y Ssy5p) y se activa luego del sensado de aminoácidos extracelulares y posiblemente intracelulares19. Ssy1 es una proteína de membrana que se asemeja a una permeasa de aminoácidos pero funciona únicamente como sensor. Ptr3 y Ssy5 interactúan con Ssy1 formando un complejo proteico dinámico. Se ha determinado un número importante de genes regulados por la actividad de este sensor y muchos de estos genes codifican para proteínas involucradas en el transporte de aminoácidos. Entre ellos, BAP2 y AGP1 responden a aminoácidos vía SPS a través de los factores de transcripción Stp1, Stp2 y Uga3520.

Los factores Stp1 y Stp2 actúan, en respuesta al sensado de aminoácidos extracelulares, sobre elementos UASaa presentes en promotores de permeasas, como por ejemplo BAP2 y BAP3. Es importante destacar la gran similitud de secuencia entre los elementos UASaa y el UASGABA21,22. Stp1 y Stp2 se sintetizan como factores latentes y permanecen retenidos en el citoplasma a través del dominio regulatorio amino terminal. También tres proteínas presentes en la membrana nuclear interna, Asi1, Asi2 y Asi3, participan en la retención de Stp1 y Stp2 latentes en el citoplasma23,24. En respuesta a aminoácidos, Ssy1 activa a la proteasa Ssy5 a que a su vez cliva el dominio regulatorio de Stp1 y de Stp2, llevando a la activación de estos factores. Sus formas procesadas son transportadas al núcleo donde transactivan genes regulados por SPS20,25-27.
Bibliografía:
1. De Virgilio, C. & Loewith, R. Cell growth control: little eukaryotes make big contributions. Oncogene 25, 6392-415 (2006).
2. Beck, T. & Hall, M. N. The TOR signalling pathway controls nuclear localization of nutrient-regulated transcription factors. Nature 402, 689-92 (1999).
3. Bermudez Moretti, M., Correa Garcia, S., Stella, C., Ramos, E. & Batlle, A. M. Delta-aminolevulinic acid transport in Saccharomyces cerevisiae. Int J Biochem 25, 1917-24 (1993).
4. Bermudez Moretti, M., Correa Garcia, S., Ramos, E. & Batlle, A. delta-Aminolevulinic acid uptake is mediated by the gamma-aminobutyric acid-specific permease UGA4. Cell Mol Biol (Noisy-le-grand) 42, 519-23 (1996).
5. Andre, B., Hein, C., Grenson, M. & Jauniaux, J. C. Cloning and expression of the UGA4 gene coding for the inducible GABA-specific transport protein of Saccharomyces cerevisiae. Mol Gen Genet 237, 17-25 (1993).
6. Uemura, T., Tomonari, Y., Kashiwagi, K. & Igarashi, K. Uptake of GABA and putrescine by UGA4 on the vacuolar membrane in Saccharomyces cerevisiae. Biochem Biophys Res Commun 315, 1082-7 (2004).
7. Andre, B. The UGA3 gene regulating the GABA catabolic pathway in Saccharomyces cerevisiae codes for a putative zinc-finger protein acting on RNA amount. Mol Gen Genet 220, 269-76 (1990).
8. Andre, B. et al. Two mutually exclusive regulatory systems inhibit UASGATA, a cluster of 5'-GAT(A/T)A-3' upstream from the UGA4 gene of Saccharomyces cerevisiae. Nucleic Acids Res 23, 558-64 (1995).
9. Talibi, D., Grenson, M. & Andre, B. Cis- and trans-acting elements determining induction of the genes of the gamma-aminobutyrate (GABA) utilization pathway in Saccharomyces cerevisiae. Nucleic Acids Res 23, 550-7 (1995).
10. Vissers, S., Andre, B., Muyldermans, F. & Grenson, M. Positive and negative regulatory elements control the expression of the UGA4 gene coding for the inducible 4-aminobutyric-acid-specific permease in Saccharomyces cerevisiae. Eur J Biochem 181, 357-61 (1989).
11. Grenson, M. 4-Aminobutyric acid (GABA) uptake in Baker's yeast Saccharomyces cerevisiae is mediated by the general amino acid permease, the proline permease and a GABA specific permease integrated into the GABA-catabolic pathway. Life Sci Adv Biochem 6, 35-39 (1987).
12. Idicula, A. M., Blatch, G. L., Cooper, T. G. & Dorrington, R. A. Binding and activation by the zinc cluster transcription factors of Saccharomyces cerevisiae. Redefining the UASGABA and its interaction with Uga3p. J Biol Chem 277, 45977-83 (2002).
13. Magasanik, B. & Kaiser, C. A. Nitrogen regulation in Saccharomyces cerevisiae. Gene 290, 1-18 (2002).
14. Kulkarni, A., Buford, T. D., Rai, R. & Cooper, T. G. Differing responses of Gat1 and Gln3 phosphorylation and localization to rapamycin and methionine sulfoximine treatment in Saccharomyces cerevisiae. FEMS Yeast Res 6, 218-29 (2006).
15. Bermudez Moretti, M. et al. Evidence that 4-aminobutyric acid and 5-aminolevulinic acid share a common transport system into Saccharomyces cerevisiae. Int J Biochem Cell Biol 27, 169-73 (1995).
16. Didion, T., Regenberg, B., Jorgensen, M. U., Kielland-Brandt, M. C. & Andersen, H. A. The permease homologue Ssy1p controls the expression of amino acid and peptide transporter genes in Saccharomyces cerevisiae. Mol Microbiol 27, 643-50 (1998).
17. Iraqui, I. et al. Amino acid signaling in Saccharomyces cerevisiae: a permease-like sensor of external amino acids and F-Box protein Grr1p are required for transcriptional induction of the AGP1 gene, which encodes a broad-specificity amino acid permease. Mol Cell Biol 19, 989-1001 (1999).
18. Klasson, H., Fink, G. R. & Ljungdahl, P. O. Ssy1p and Ptr3p are plasma membrane components of a yeast system that senses extracellular amino acids. Mol Cell Biol 19, 5405-16 (1999).
19. Wu, B. et al. Competitive intra- and extracellular nutrient sensing by the transporter homologue Ssy1p. J Cell Biol 173, 327-31 (2006).
20. Abdel-Sater, F., Iraqui, I., Urrestarazu, A. & Andre, B. The external amino acid signaling pathway promotes activation of Stp1 and Uga35/Dal81 transcription factors for induction of the AGP1 gene in Saccharomyces cerevisiae. Genetics 166, 1727-39 (2004).
21. de Boer, M. et al. Stp1p, Stp2p and Abf1p are involved in regulation of expression of the amino acid transporter gene BAP3 of Saccharomyces cerevisiae. Nucleic Acids Res 28, 974-81 (2000).
22. Nielsen, P. S. et al. Transcriptional regulation of the Saccharomyces cerevisiae amino acid permease gene BAP2. Mol Gen Genet 264, 613-22 (2001).
23. Boban, M. & Ljungdahl, P. O. Dal81 enhances Stp1- and Stp2-dependent transcription necessitating negative modulation by inner nuclear membrane protein Asi1 in Saccharomyces cerevisiae. Genetics 176, 2087-97 (2007).
24. Boban, M. et al. Asi1 is an inner nuclear membrane protein that restricts promoter access of two latent transcription factors. J Cell Biol 173, 695-707 (2006).
25. Abdel-Sater, F., El Bakkoury, M., Urrestarazu, A., Vissers, S. & Andre, B. Amino acid signaling in yeast: casein kinase I and the Ssy5 endoprotease are key determinants of endoproteolytic activation of the membrane-bound Stp1 transcription factor. Mol Cell Biol 24, 9771-85 (2004).
26. Andreasson, C., Heessen, S. & Ljungdahl, P. O. Regulation of transcription factor latency by receptor-activated proteolysis. Genes Dev 20, 1563-8 (2006).
27. Andreasson, C. & Ljungdahl, P. O. The N-terminal regulatory domain of Stp1p is modular and, fused to an artificial transcription factor, confers full Ssy1p-Ptr3p-Ssy5p sensor control. Mol Cell Biol 24, 7503-13 (2004).
lunes, 28 de julio de 2008
Decimoprimer semana (24/07/08)
Le preguntamos nuestras dudas a Sabrina, ella nos dio papers, nos explico otros temas y nos dio más información.
jueves, 17 de julio de 2008
Décima semana (17/07/08)
Medio LB (para crecer bacterias):
·Extracto de levadura 0,5%
·NaCl 1%
·Peptona 1%
·Agar 2%
Medio YPD (medio rico, para crecer levaduras)
·Y (extracto de levadura) 1%
·P (peptona) 2%
·D (dextrosa/glucosa) 2%
·Agar 2%
Medio YNB-DO (medio mínimo URA(-) para crecer levaduras)
·YNB (yeast nitrogen base) 0,67%
·Glucosa 2%
·Drop out URA(-) 0,19 %
·Agar 2,5%
A todos los medios les agregamos agar porque vamos a hacer placas.
Autoclavamos los medios junto con tips y erlenmeyers para esterilizarlos.
Preparamos 15 placas con los medios, 5 placas para cada uno y las rotulamos.
Preparamos un buffer que hacía falta en el laboratorio, buffer Z usado, entre otras cosas, para el ensayo de B-gal de hace dos semanas.
Pesamos sus componentes al igual que hicimos con los medios, pero sin autoclavar.
Susana nos explicó como se construyen los primers y nos dio un problema para hacer.
Novena semana (10/07/08)
 Comprobamos en qué nos habíamos equivocado la vez pasada, y tras pasar todo de nuevo al Excel pudimos darnos cuenta que el error que había era de unos paréntesis en las ecuaciones para sacar las unidades miller.
Comprobamos en qué nos habíamos equivocado la vez pasada, y tras pasar todo de nuevo al Excel pudimos darnos cuenta que el error que había era de unos paréntesis en las ecuaciones para sacar las unidades miller. Ésta técnica consiste básicamente en:
· fijar o dejar todo estático (uniendo covalentemente si es que hay, la unión con la proteína) utilizando formaldehido
· luego sonicar para partir el ADN en fragmentos más chicos
· se agrega una proteína que ayuda a limpiar las proteínas que están de más
· se le pone un anticuerpo que se pega a la proteína que me interesa
· luego se agrega una bola de proteína que también se pega al anticuerpo y todo esto precipita
· se hace PCR y se analiza el contenido.
miércoles, 9 de julio de 2008
Octava semana (03/07/08)
Se le agrega un sustrato a las muestras que contienen otros compuestos, para saber si estas interfieren en la actividad de la enzima, el producto que da esta al reaccionar con ONPG es amarillo y se mide en espectrofotómetro.
Tomamos una alícuota de los tubos de la semana pasada y les agregamos Buffer Z. Colocamos SDS y cloroformo para que luego al poner ONPG, esta sustancia pueda entrar, al agregar esto, la b-galactosidasa comienza a catalizar la reacción y cuando haya producto (este amarillo) se la frena con Na2CO3 y se lo coloca en frió. (hacemos las muestras por duplicado y cada 30 segundos agregamos ONPG a los tubos).

Anotamos el tiempo inicial y el tiempo final de la reacción. Sacamos la densidad celular de los ml sobrantes de los tubos a una Absorbancia a 570 nm. La Absorbancia para las unidades Miller es a 420 nm. Hacemos una tabla que incluya todos los valores y además un gráfico donde figure las unidades Miller en función del tiempo.
sábado, 28 de junio de 2008
Séptima semana (26/06/08)
Día 1: se toma una colonia de levadura que contienen un plásmido ( YEP-357) y se pone en un medio mínimo, se lo deja toda la noche.
Día 2: se hace una dilución del medio y se lo deja toda la noche.
Día 3: el medio se divide en tres tubos que se centrifugan quedándonos con el pellet y agregándole de nuevo el medio mínimo pero fresco (a uno de los tres tubos se le agrega Leucina (un aminoácido)). Después de 30 minutos al tubo de los aminoácidos y a otro se le agrega GABA.
Se toma una muestra a tiempo 0 de los tres tubos y otras a los 120 minutos.
El objetivo es saber en que condiciones el gen se induce mejor, si afecta la presencia de aminoácidos en la inducción y como se ven estos cambios según la variación del tiempo.
Sabrina también nos explicó como hacer para saber si una proteína interactúa con el gen UGA 4. para eso se usa formaldehido que une covalentemente a la proteína con el gen. Después un anticuerpo y una bola de agarosa se unen con la proteína. Ésto se hace precipitar con ayuda de la centrífuga.
Luego de esto puede haber 2 posibilidades: que la proteína se pegue a UGA 4 y precipite con este, o que se pegue a otra cosa y quede el gen UGA 4 en el sobrenadante.
Si queda UGA 4 en el sobrenadante significa que la proteína no interactúa con este.
Se verifica con PCR
miércoles, 25 de junio de 2008
Sexta semana (19/06/2008)
Luego Sabrina nos explicó lo que ibamos a hacer la próxima semana.
viernes, 13 de junio de 2008
Quinta semana (12/06/08)
Preparamos un medio de crecimiento, con:
Extractos de levadura 1%
Peptona 2%
Glucosa 2%
Agar 2% (para preparar placas).
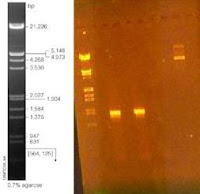
en el primer pocillo esta el marker y pocillo de por medio esta las PCR primero las positivas y luego la negativa (como no hay nada podemos afirmar que no hubo contaminación), y la ultima es la del plásmido.
la persona que vende el marker entrega también con éste un papel mostrando como va a correr y las bandas que indican tamaños según los pares de bases. Así podemos saber cuantos pares de bases tienen las PCR y el plásmido.
lunes, 9 de junio de 2008
Cuarta semana (05/06/08)
Tomamos una alícuota de 5 ml de nuestro cultivo de bacterias E.Coli y lo centrifugamos, quedándonos con el pellet (el sobrenadante esta compuesto por los componentes del medio y en el pellet estan las bacterias)
Agregamos un Buffer que tiene EDTA (por su acción quelante ayuda a desestabilizar más adelante a la membrana bacteriana) también tiene un colorante azul, Lyse Blue, (que me indica cuando todo esta homogeneizado).
Agregamos otra solución Buffer (compuesta por NaOH y SDS). Se produce una lisis básica, y el colorante de transparente cambia a azul. Hay que mezclar por inmersión, ya que todo lo que tengo dentro del tubo es demasiado sensible, hasta que el color azul sea igual en todos lados.
Agregamos el tercer Buffer que baja el pH favoreciendo la renaturalización del ADN. pero como es un cambio brusco se renaturaliza mal y lo que obtengo es todo un precipitado de restos de membranas,paredes ADN genómico etc., el colorante pasa de azul a incoloro (se mezcla por inmersión nuevamente).
Como el plásmido es muy chiquito y además esta unido fisicamente, no se separa y se puede renaturalizar bien. Me queda en solución.
centrifugamos.
Colocamos la solución en una columnita donde va a quedar unido en la recina el ADN, pasando a través de ella (enzimas, etc). Centrifugar.
Lavamos con un Buffer que separa proteínas y sales del plásmido. Centrifugar.
Se le agrega otro Buffer con el fin de seguir lavando y purificando nuestro plásmido. Centrifugamos. Colocamos otro Buffer que va a despegar el plásmido de la columnita (centrifugando para asegurarnos que bajo todo el plásmido purificado).
Preparamos un Gel de agarosa 1% p/v en 45 ml
Colocamos en los pocillos del gel las tres PCR y la solución del plásmido (cada una con 1,25 μl de Buffer loading (que por su densidad, contiene glicerina, nos ayuda a sembrar haciendo lo que sembramos baje al posillo y no se quede flotando, y el colorante que tiene nos permite visualizar el frente de corrido).
La semana que viene vamos a ver los resultados de otro gel de agarosa.
sábado, 7 de junio de 2008
Tercer semana (29/05/08)
Buffer
dntp
MgCl2
Primer
DNA pol (Taq polimerasa, termoestable, sobrevive a las altas temperaturas)
Molde de DNA genómico
H2O
Calculamos sus concentraciones necesarias para que todo este en 150 μl
*A la hora de mezclar todo es importante tener en cuenta que lo primero que se debe poner es el agua y el Buffer porque los demás componentes son muy sensibles como para colocarlos en el tubo vació sin que nada les pase.
Preparamos tres tubos de PCR con 50 μl cada uno de mezcla. Dos de ellos con el molde (los llamaremos positivos (+)) y uno sin él (a este lo llamaremos negativo (-)). Es necesario hacer el tubo negativo porque nos indica si hubo contaminación o no y si lo que tengo en los tubos es sólo el ADN gel gen UGA-3 o también otras cosas. Nos queremos asegurar de estar amplificando sólo la secuencia seleccionada.
Para hacer la PCR se hace un programa dónde se calienta todo a 94°C (para separar las cadenas de ADN) se baja la temperatura a 63°C (a esta temperatura se unen los primers) y luego se sube a 72°C (que es la temperatura óptima para la polimerización). Este programa se hace en ciclos para amplificarlo y tener mucho producto.
94°C 5min
94°C 1min\
63°C 1min ) se repite 30 veces.
72°C 2min/
72°C 5min
domingo, 25 de mayo de 2008
Segunda semana (22/05/08)
 Sembramos bacterias E. Coli con un plásmido dentro, con el fin de multiplicar masivamente su número y así poder tener muchos plásmidos que luego utilizaremos para un clonado.
Sembramos bacterias E. Coli con un plásmido dentro, con el fin de multiplicar masivamente su número y así poder tener muchos plásmidos que luego utilizaremos para un clonado.Usamos este plásmido porque se puede replicar en levaduras y en bacterias y además posee un gen reportero (β-Galactosa).
jueves, 22 de mayo de 2008
Primer semana (15/05/08)
Luego a las 10 de la mañana nos retiramos para presenciar una charla instructiva que era proveída por la UBA. Ésta estaba compuesta por dos videos y explicaba y nombraba las normas de seguridad y en que uno debe trabajar y qué debemos hacer en casos de emergencias.
Al finalizar la charla volvimos al laboratorio y Sabrina sola nos siguió instruyendo sobre contenidos teóricos, nos explicó los temas más a fondo y también nos nombró unas técnicas que utilizaríamos a lo largo de la pasantía (como PCR), nos mostró el funcionamiento de algunos equipos de trabajo y algunos materiales. También nos mandó información del tema vía internet.
Algunos temas que tratamos:
Bacteria: E. Coli
Gen: UGA4 de Saccharomyces cerevisiae
Técnica de clonado: PCR
Iván y Victoria
jueves, 8 de mayo de 2008
Proyecto Química ORT del Genoma Humano
Por cuarto año consecutivo los alumnos del ultimo año de la especialidad química de la escuela técnica ORT (Argentina) realizarán sus proyectos finales en distintas universidades y centros de investigación con profesionales reconocidos. La metodología de trabajo evita la transposición didáctica permitiendo a los estudiantes realizar su actividad final en los lugares donde se está generando el conocimiento.
La articulación entre el nivel medio pos grado universitario es auspiciada por el CONICET(Consejo Nacional de Investigación Científica y Técnica.
Los proyectos de investigación para el año en curso son:
1. Evaluación de la expresión de la molecula de adhesión cadherina epitelial en tejidos humanos normales y tumorales.
Investigador: Dra. Mónica Vazquez-Levin.
Instituto: Instituto de Biología y Medicina Molecular (IBYME - CONICET).
Alumnos: Yael Dobzewicz Y Gala Szapiro.
Link: http://www.proyecto-6q.blogspot.com/
2. Acción del hexaclorobenceno (HCB) sobre la uroporfirinogeno de una línea celular hepatocitos humanos. Mecanismo de acción.
Investigador: Dra. María del Carmen Ríos de Molina.
Instituto: Departamento Química Biológica, Facultad de Ciencias Exactas y Naturales.
Alumnos: Lucas Toiw y Uriel Frid.
Link: http://www.proyectofrid-toiw08.blogspot.com/
3. Modelos experimentales de enfermedades metabólicas: Porfiria y Síndrome Metabólico. Proteómica y Metabolómica de estos disturbios.
Investigador: Dra. Marta Blanca Mazzetti.
Instituto: Departamento de Química Biológica, Facultad de Ciencias Exactas y Naturales (UBA).
Alumnos: Maria Duperron y Mauro Elencwajg.
Link: http://proyectofinal6q.blogspot.com/
4. Factores no hemodinámicos relacionados con la génesis y evolución de la proteinuria durante la enfermedad renal progresiva.
Investigador: Dra. Elsa Zotta.
Instituto: Laboratorio de Fisiopatogénia, Departamento de fisiología, Facultad de medicina, UBA.
Link: http://www.proyectoq08.blogpsot.com/
5. Estudio de la participacion de la anandamida en la regulacion de la interaccion espermatozoide-ovioducto en un modelo bovino.
Investigador: Dra. Silvina Perez Martinez.
Alumnos: Judith Arenas Tenenbaum y Melina Braverman
Link: http://www.scienceproject08.blogspot.com/
6. El estudio de los mecanismos moleculares involucrados en la regulación del gen UGA4 de Saccharomyces cervisiae en respuesta a cambios en la disponibilidad de nutrientes con el fin de dilucidar las distintas cascadas de señales desencadenadas por dichos nutrientes y establecer sus interconexiones.
Investigador: Dra Susana Correa García.
Instituto: Departamento de Química Biologica, Facultad de ciencias Exactas y Naturales, Universidad de Buenos Aires.
Alumnos: Iván Mikiej y Victoria Salama
Link: http://www.proyectoquimica22.blogspot.com/
7. Diagnóstico de la enfermedad de Von Willebrand (VWD) tipo 2N por técnicas fenotípicas y genotípicas.
Investigador: Dra. Adriana I. Woods.
Instituto: Instituto de Investigaciones Hematológicas "Mariano R. Castex" de la Academia Nacional de Medicina.
Alumnos: Abigail Skverer y Daniela Lin
Link: http://www.vwd-diagnostico.blogspot.com/
8. Estudio de la mielinogenesis en el sistema nervioso periferico en condiciones fisiologicas y patologicas. Participacion de celulas pluripotentes en el proceso de degeneracion-regeneracion nerviosa.
Investigador: Dra Patricia Setton-Avruj.
Instituto: Dpto de Quimica Biologica, Facultad de Farmacia y Bioquimica (IQUIFIB-UBA-CONICET).
Alumnos: Averbuj Daniel y Eitan Rozenszajn.
Link: http://www.proyectoaverbuj-rozenszajn.blogspot.com/
9. Diseño y desarrollo de vacunas antitumorales empleando bacterias y celulas tumorales modificadas con genes inmunomodiladores. Estudio de los mecanismos inmunes inducidos.
Investigador: Claudia I. Waldner y Claudia Mongini.
Instituto: Laboratorio de inmunologia celular y molecular. Centro de Estudios Farmacologicos y Botanicos (CEFYBO. CONICET-UBA)
Alumnos: Amalia Surijon y Maria Belen Tolava Rivero
Link: http://www.amibeluproyecto.blogspot.com/
10. Marcadores Geneticos Asociados al Cáncer
Investigador: Dr.Javier Hernán Cotignola
Instituto: Laboratorio de Cáncer y Apoptosis del Departamento de Quimica Biologica de la Facultad de Ciencias Exactas y Naturales de la Universidad de Buenos Aires.
Alumnos: Ayelén Marano y Solange Perchik
Link: http://marcadoresgeneticos.blogspot.com/
11. Expresion del canal de la conductancia transmembranal de la Fibrosis Quistica (CFTR) en placentas Preeclampticas: posible rol en la regulacion de la actividad de la Acuoporina-9 (AQP9).
Investigador: Dr Alicia E Damiano.
Instituto: Catedra de Biologia Celular, Departamento de Ciencias Biologicas, Facultad de Farmacia y Bioquimica (UBA)
Alumnos: Gabriel Colodenco y Martín Sapir.
Link: http://www.proyectofinalcs.blogspot.com/
12. Proteómica de factores secretados por células de cáncer de mama y mama normal con capacidad inhibitoria de la producción lipídica.
Investigador: Dra.Guerra de Grignoli Liliana Noemi
Instituto: Departamento de Ciencias biologicas. Facultad de ciencias exactas y naturales,UBA-CONICET.
Alumnos: Fabiana Durante y Tamara Broitman.
Link: http://tyf-proyecto08.blogspot.com/
13. Neovascularizaciòn en un modelo murino de inflamaciòn aguda inducida por LPS.
Investigador: Eulalia de la Torre.
Instituto: Facultad de Medicina - Uba - Laboratorio de Inmunofarmacologia
Alumnos: Federico Mauas Walach, Pablo Kuleff.
Link: http://finalproyectort.blogspot.com/
14. Interacción de sumo-1 y mage-a2 en la regulacion del oncosupresor p53
Investigador: Martín Monte
Instituto: Facultad de Ciencias Exactas y Naturales (UBA)
Alumnos: Leonel Stermann y Yair Litman
Link: http://proyectosumo.blogspot.com/
15. Estudio de la Growth Associated Protein (GAP-43), su interacción con la Ubicutina y su participación en el control del ciclo celular en células NIH3T3 transfectadas en forma estable y transiente. Efecto de la Apo-transferrina en la remielización: participación de la vía Notch en la diferenciación oligodendrioglial.
Investigador: Doctora Ana M. Adamo.
Instituto: Departamento de Química Biológica Patológica, Facultad de Farmacia y Bioquímica de la UBA. CONICET.
Alumnos: Rodrigo C. Pampin y Robby Mattes
Link: http://www.gap-43.blogspot.com/
jueves, 3 de abril de 2008
Presentacion
Este es el blog de nuestro proyecto final, de la especialidad de Química.
La investigadora: Dra Susana Correa García.