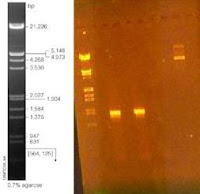
Hicimos una inducción.
Día 1: se toma una colonia de levadura que contienen un plásmido ( YEP-357) y se pone en un medio mínimo, se lo deja toda la noche.
Día 2: se hace una dilución del medio y se lo deja toda la noche.
Día 3: el medio se divide en tres tubos que se centrifugan quedándonos con el pellet y agregándole de nuevo el medio mínimo pero fresco (a uno de los tres tubos se le agrega Leucina (un aminoácido)). Después de 30 minutos al tubo de los aminoácidos y a otro se le agrega GABA.
Se toma una muestra a tiempo 0 de los tres tubos y otras a los 120 minutos.
El objetivo es saber en que condiciones el gen se induce mejor, si afecta la presencia de aminoácidos en la inducción y como se ven estos cambios según la variación del tiempo.
Sabrina también nos explicó como hacer para saber si una proteína interactúa con el gen UGA 4. para eso se usa formaldehido que une covalentemente a la proteína con el gen. Después un anticuerpo y una bola de agarosa se unen con la proteína. Ésto se hace precipitar con ayuda de la centrífuga.
Luego de esto puede haber 2 posibilidades: que la proteína se pegue a UGA 4 y precipite con este, o que se pegue a otra cosa y quede el gen UGA 4 en el sobrenadante.
Si queda UGA 4 en el sobrenadante significa que la proteína no interactúa con este.
Se verifica con PCR
Día 1: se toma una colonia de levadura que contienen un plásmido ( YEP-357) y se pone en un medio mínimo, se lo deja toda la noche.
Día 2: se hace una dilución del medio y se lo deja toda la noche.
Día 3: el medio se divide en tres tubos que se centrifugan quedándonos con el pellet y agregándole de nuevo el medio mínimo pero fresco (a uno de los tres tubos se le agrega Leucina (un aminoácido)). Después de 30 minutos al tubo de los aminoácidos y a otro se le agrega GABA.
Se toma una muestra a tiempo 0 de los tres tubos y otras a los 120 minutos.
El objetivo es saber en que condiciones el gen se induce mejor, si afecta la presencia de aminoácidos en la inducción y como se ven estos cambios según la variación del tiempo.
Sabrina también nos explicó como hacer para saber si una proteína interactúa con el gen UGA 4. para eso se usa formaldehido que une covalentemente a la proteína con el gen. Después un anticuerpo y una bola de agarosa se unen con la proteína. Ésto se hace precipitar con ayuda de la centrífuga.
Luego de esto puede haber 2 posibilidades: que la proteína se pegue a UGA 4 y precipite con este, o que se pegue a otra cosa y quede el gen UGA 4 en el sobrenadante.
Si queda UGA 4 en el sobrenadante significa que la proteína no interactúa con este.
Se verifica con PCR